Molegro Virtual Docker注册破解版(MVD)破解版是一个用于研究和预测配体如何与大分子相互作用的集成环境。通过迭代评估许多候选溶液(配体构象)并估计它们与大分子相互作用的能量来完成配体结合模式的鉴定。返回得分最高的解决方案以进行进一步分析。 MVD需要蛋白质和配体的三维结构(通常衍生自X射线/ NMR实验或同源建模)。 MVD执行灵活的配体对接,因此在对接期间将确定配体的最佳几何形状。。 Molegro Virtual Docker处理对接过程的所有方面,从分子的制备到确定靶蛋白的潜在结合位点,以及预测配体的结合模式。软件基于新颖的优化技术和专注于可用性和生产力的用户界面体验,提供高质量的对接。可提供高对接精度,已证明对接引擎能够以高精度正确识别绑定模式。在识别正确的绑定模式方面,Molegro Virtual Docker已经证明胜过其他对接程序。它还带有一个易于使用的界面,内置向导使用户可以轻松设置和执行对接运行。提供先进的可视化和分析工具来检查配体 - 受体相互作用并微调发现的对接解决方案。本次小编带来的是最新2013破解版,含注册机,亲测可完美激活软件,有需要的朋友不要错过了!
安装破解教程
1、在本站下载并解压,得到MVD2013_6.0.1.exe安装程序和Crack破解文件夹

2、双击MVD2013_6.0.1.exe运行安装,点击next
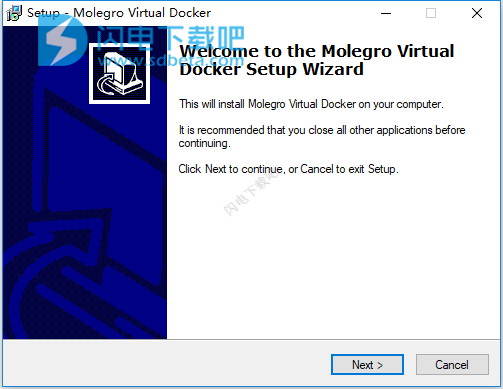
3、勾选我接受协议,点击next
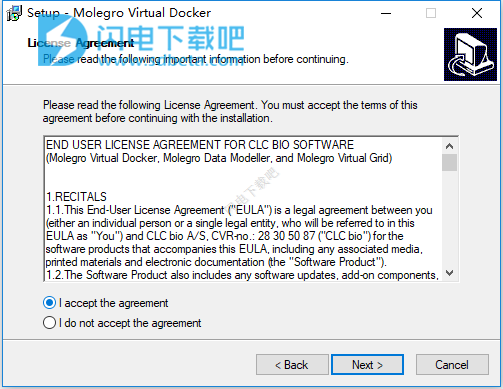
4、点击浏览选择软件安装路径,点击next
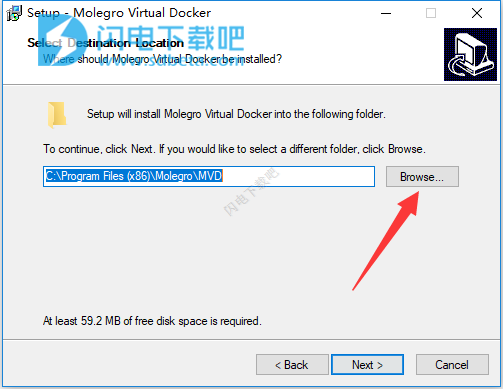
5、点击install安装
6、安装中,大家稍等一会儿
7、安装完成,点击finish退出向导
8、将crack破解文件夹中的Keygen.jar复制到安装目录中bin文件夹下,运行Keygen.jar,输入用户名和组织,点击生成许可证!
9、
把生成的许可证复制到安装目录的bin文件夹
Molegro Virtual Docker软件功能
1、高准确性的docking结果,与其他docking软件想比较之下,为准确率最好的(MVD: 87%, Glide: 82%, Surflex: 75%, FlexX: 58%)
2、简单易用的软件界面:提供互动步骤方法 (wizard),让使用者可以很快的设定及执行docking,也针对docking结果提供完整的视觉及分析工具。
3、跨平台的整合:Molegro Virtual Docker 提供在Linux, Windows,及Mac平台的版本以及其结果可以在各平台存取
Molegro Virtual Docker软件特色
1、高精度对接:已经得到验证,能够高精度地正确识别结合结合模式。
2、功能全面:包括诱导契合对接(侧链柔性),基于结构相似性的虚拟筛选,柔性的小分子叠合,用人工神经网络或MLR建立回归模型。
3、高级的视图与分析工具:可以用来分析配体-受体相互作用以及精细调节以观察对接结果
4、网格计算:用Molegro Virtual Grid在多个机器上实现分布式对接计算
5、跨平台:支持Windows,Mac与Linux
Molegro Data Modeller软件功能
1、可视化
不同的可视化类型拥有高度的互动性。电子表格中的选择被直接显示在图表中,反之亦然。它也可以应用各种用户定义的着色方案和应用抖动(添加人工噪声到数据图 ).它也能让高维数据形象化。使用内置的弹簧质量绘图模型,高维数据能投影成2D或者3D。
2、化学
Molegro Data Modeller支持化学数据:MDM能理解SMILES和SDF文件,也能在电子表格中或者二维绘图仪中创建2D图。
3、聚类
Molegro Data Modeller能进行不同类型的聚类:K表示聚类和基于门限值的聚类(这两种效率都非常高),以及一个基于密度的聚类方案(它能获取更复杂的集群形态)。
4、主成分分析(PCA)
主成分分析是减少数据集维度的一种方法。一组新的主要成分是通过原始描述符的线性组合创建的。然后只需通过保持描述符对方差的最大贡献来减少描述符的数量。
5、代数数据转换
在栏目中直接进行代数变换也是可能的:比如"New Activity = log(Act) + Beta^2"将在这个表述的基础上创建一个新栏目。
6、高级子集的创建
Molegro Data Modeller提供了一种基于网格的方法,用于创建一个数据集的多个子集
7、跨平台(Cross-Platform)
Molegro Data Modeller可以在以下系统里运行:
Windows XP, Vista, and 7.
Mac OS X (10.4 and later, PowerPC and Intel supported).
大部分主要的Linux配置。
Molegro Data Modeller软件特色
1、回归和分类- Molegro Data Modeller提供不同种类的数据模型;
2、特征选择和交叉验证-特征选择可以容易地通过回归向导来设置;
3、交叉验证使用的是指定数目的随机褶皱(可以使用给一法Leave-One-Out或手动创建褶皱)。
使用帮助
1、导入和制备分子
为了使MVD能够以最佳方式发挥作用,必须在对接开始之前准确地准备工作区中的分子。分子可以在MVD内部制备,也可以在外部由另一个程序制备(例如来自CCG [CCG]的MOE或来自Schrodinger,LLC的Maestro)
[SCHRODINGER])。在本教程中,我们将使用MVD中提供的内置准备方法。
2、文件导入
如果工作空间不为空,请先将其清除(选择File I ClearWorkspace)。接下来我们将添加一些结构。这可以通过选择File |来完成导入分子......或通过拖放分子结构文件。 MVD支持PDB,Mol2,SDF及其自己的基于XML的格式MVDML。
首先导入文件1HVR。 pdb来自安装示例目录(位于MVD安装文件夹中)。该文件(与XK263复合的HIV-1蛋白酶)是来自RCSB蛋白质数据库的未修改文件。
(www.pdb.org)。选择要导入的分子
出现“导入分子”对话框(参见图2)。
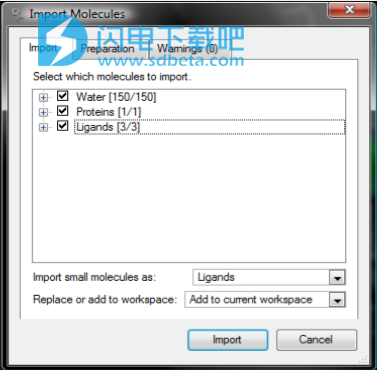
图2:从PDB文件导入1HVR。
取消选择辅助因子,因为在这个例子中我们不需要这些。 导入对话框显示两种蛋白质:实际上这些是来自同一蛋白质的两条链。 它还表明在PDB文件中检测到配体。
3、选择准备类型
选择Preparation选项卡(参见图3)。 一些结构包含有关债券类型和债券订单的信息,并且分配了明确的氢。 但是,PDB文件通常具有较差或缺少明确氢的分配,并且PDB文件格式不能容纳债券订单信息。 将所有下方全部设置为始终。 这确保了所有准备工作都将通过MVD完成。
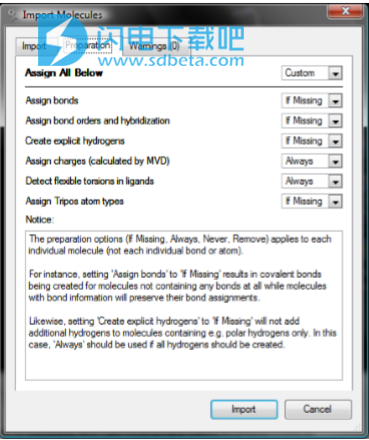
图3:准备PDB文件。
如果预先准备好蛋白质结构并以能够处理所有结构信息(例如Mol2文件格式)的格式保存,则应通过默认的准备设置If Missing导入。如果在文件中找不到所需信息,则此设置仅执行给定准备。
注意:默认情况下,忽略分子的任何电荷信息(“分配电荷=始终”表示MVD的内部电荷方案始终用于计算电荷)。如果要使用分子存储的部分电荷,请设置'分配电荷=如果失踪 - 这样MVD将仅计算没有电荷信息的分子的电荷。如果从分子输入电荷(例如,提供Mol2文件),分配给氢的部分电荷将被移动到键合的重原子,因为明确的氢是在对接过程中使用的评分功能没有考虑到。
4、检查警告导
入对话框中的最后一个选项卡(警告(0))显示结构文件的潜在问题。在这种情况下,不会报告任何警告。
现在单击“导入”按钮。 蛋白质和配体出现在可视化窗口中(参见图4)。
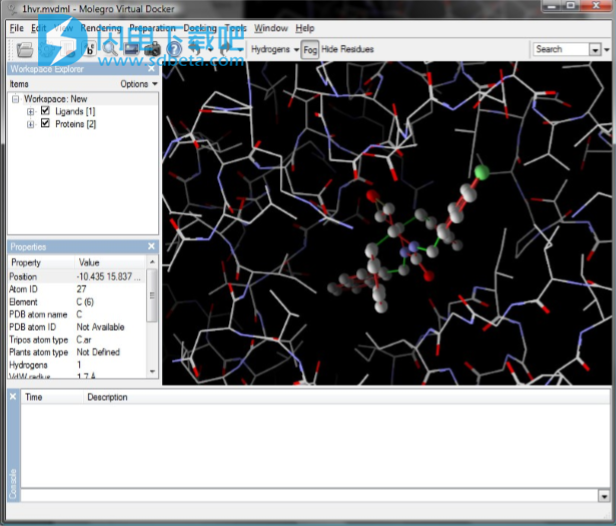
图4:导入的结构。
为了检查导入的配体,通过单击Workspace Explorer中Proteins类别旁边的复选框隐藏蛋白质(参见图5)。
图5:隐藏蛋白质。
现在放大配体(参见图6)。
可以使用以下任一方式执行缩放:
。 鼠标滚轮
。 按住两个鼠标按钮
。 按shift并按住鼠标左键也可以从Workspace Explorer中的配体上下文菜单中选择Fit to Screen。 注意,配体已被指定为键序,已检测到芳环,并且已添加显性氢。 还要注意一些债券是绿色的。 在对接模拟期间,这些键将被设置为灵活的。 如果在模拟过程中粘合应保持刚性,请右键单击它,然后从上下文菜单中选择“设置柔性”。
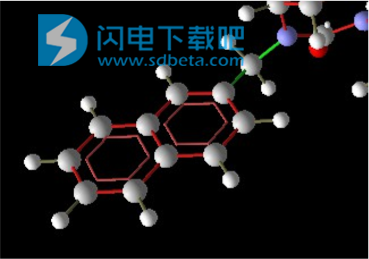
图6:检查灵活的债券。
添加分子表面接下来,我们将添加表面以获得蛋白质结构的印象。
我们将通过从Workspace Explorer的Proteins上下文菜单中选择Create Surface .. ...来完成此操作(参见图7)。
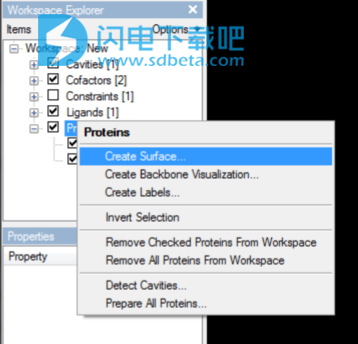
图7:表面创建。
在出现的对话框中,只需单击“确定”。 这将基于默认设置产生蛋白质表面,所述默认设置是根据静电势着色的不透明溶剂可接触表面(红色和蓝色区域对应于分别具有带负电和带正电荷的残基的区域)。 请注意,曲面也显示为Workspace Explorer(Surfaces类别)中的元素。
5、预测结合位点
接下来,我们将尝试缩小蛋白质的潜在结合位点。 这可以通过选择Preparation |来自动完成 检测空腔。 按下OK按钮后,系统将使用附录IV:腔体预测中描述的算法预测蛋白质中心的结合位点(参见图8)。

图8:预测的结合位点。
现在我们准备开始对接了。 要更清楚地了解此过程,请先选择View I Reset View !. 这将重置3D视图(并隐藏曲面)。 现在选择View | 停靠视图。 这将切换到配置和姿势具有不同颜色和加盖棒表示而不是球棒的视图。
















