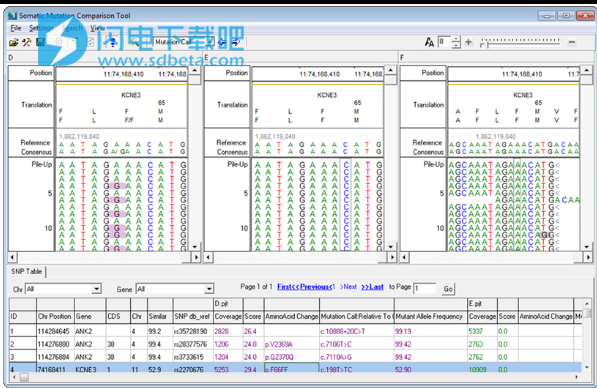
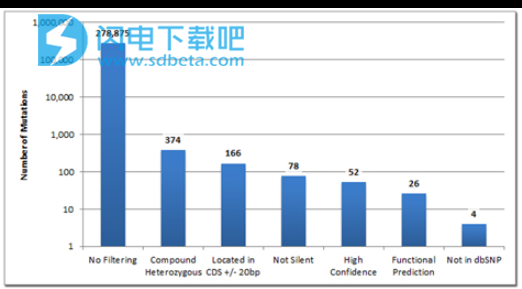
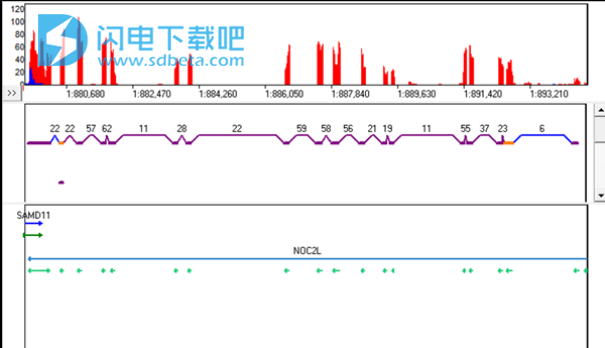
在这个6个家庭中,有两个受影响的孩子,整个家庭的外显子组,母亲,父亲,两个正常以及两个受影响的孩子进行了测序。NextGENe的变异比较工具迅速减少了近280,000个共享变体,使遗传学家能够快速确定潜在的致病突变。
SoftGenetics已经为NextGENe软件开发了一种改进的Burrows-Wheeler变换(BWT)比对方法,该方法包括对其他方法的几项改进,以便以高精度和高速度快速准确地将序列读数整合到整个大基因组参考(如人类基因组)。
NextGENe的全基因组比对算法是一个多步骤过程,首先对齐完全匹配的读数,然后用用户设定的错配比例进行读取,最后将剩余的读数分解为种子。然后,算法通过匹配小于读取长度的种子将读取与大型基因组对齐,然后扩展比对以找到整个读取的最佳匹配位置。这允许使用indel对齐长读取和读取。
Burrows-Wheeler变换是一种广泛接受的数据压缩算法,自1994年以来一直在使用。大规模并行序列发生器,如Ion Torrent Proton™,Illumina GA,HiSeq和MiSeq; 罗氏FLX和Junior以及Applied BioSystems SOLiD系统平台每次运行能够产生数百万或数十亿的读数,这导致人们对使用BWT算法将大量样本读数与整个基因组对齐感兴趣。该算法已经通过诸如Bowtie之类的对齐程序成功地用于此目的。2009年5月,来自Wellcome Trust Sanger Institute的研究人员发表了一篇论文,详细介绍了他们的新型BWT校准方法BWA,
为了有效地比对大基因组并定位第二代测序数据中发现的Indel,NextGENe使用改进的BWA方法。以下带注释的预索引参考基因组与NextGENe软件用户互补。可根据要求提供额外的注释基因组。
下一代RNA测序是检测整个转录组的有力工具,但是当将RNA-seq数据与DNA序列比对时未遇到的参考序列比对时,可变剪接和融合基因存在挑战。外显子内的短读取通常能够正常对齐,但跨越外显子 - 外显子连接和融合基因的读数会产生挑战。NextGENe Software利用分析工具为Illoi GA,HiSeq和MiSeq系统,Roche / 454 GS FLX,FLX Titanium和Junior,Life Technologies SOLiD系统以及Ion Torrent PGM和Proton的测序数据提供分析工具,为这些问题提供解决方案系统。利用Q-PALMA,Supersplat和TopHat等程序中没有的新算法,NextGENe可以识别属于注释和新颖脚本的读取,同时提供不需要使用脚本或命令行的高度图形界面的附加好处。变异检测,表达水平分析,
NextGENe的RNA-seq分析方法利用了四步专有算法,确保跨越外显子连接的读数对齐。该方法使用先前已知的同种型剪接位点,但仍允许检测新的转录物。使用NextGENe的全基因组比对方法将读数与预先索引的参考对齐。基于比对预测转录物。将这些预测与已知的转录物进行比较,并用于产生特异于样品的转录组参考。最后,将原始读数与发现的转录物对齐以确保最佳比对,回忆已发现的转录物并对SNP和短插入物进行突变检测。将这些预测与已知的转录物进行比较,并用于产生特异于样品的转录组参考。最后,将原始读数与发现的转录物对齐以确保最佳比对,回忆已发现的转录物并对SNP和短插入物进行突变检测。将这些预测与已知的转录物进行比较,并用于产生特异于样品的转录组参考。最后,将原始读数与发现的转录物对齐以确保最佳比对,回忆已发现的转录物并对SNP和短插入物进行突变检测。
图4:突变报告
十、表达分析
miRNA,ChIP-Seq,SAGE和DGE分析
NextGENe软件包括用于分析小RNA,(microRNA),ChIP-Seq,SAGE以及来自Ion Torrent,Illumina GA,Hi-Seq和MiSeq平台,Applied Biosystems的SOLiD系统和Roche GS FLX的数据的DGE分析的独特工具, FLX Titanium或Junior系统。NextGENe Viewer为生物学家提供了详细查看结果的能力。CLC bio,DNASTARSeqManNGEN®或MAQ&SOAP,Top Hat,BWA和Bowtie等学术工具等程序无法实现高水平的可视化。用户还可以使用查看器中的峰识别工具手动调整峰值检测设置。或MAQ&SOAP,Top Hat,BWA和Bowtie等学术工具。用户还可以使用查看器中的峰识别工具手动调整峰值检测设置。或MAQ&SOAP,Top Hat,BWA和Bowtie等学术工具。用户还可以使用查看器中的峰识别工具手动调整峰值检测设置。
可以将miRNA测序数据与miRBase参考比对以检测已知miRNA的存在,报告每种miRNA序列的表达水平。或者,可以使用NextGENe的峰识别工具将任何小RNA样本数据集映射到基因组参考上以检测新的小RNA。
NextGENe还包括专门用于处理染色质免疫沉淀DNA测序数据(ChIP-Seq)的软件工具 。这些工具的工作原理是将样本数据与参考序列对齐,利用峰值检测覆盖率并提供专门的峰值报告,其中包括每个峰的详细信息。
对齐和初始峰检测完成后,结果将显示在NextGENe Viewer中,并突出显示峰。NextGENe Viewer为生物学家提供了详细查看结果的能力
NextGENe软件充分利用了短序列读数,并具有分析SAGE的工具 标签。SAGE库可用,其中包含与特定基因相关的序列标签列表。NextGENe可以加载这些库作为参考,并将序列读数与适当的序列标签对齐。标签库的对齐仅在序列的正向方向上执行,不实现反向互补。创建数字基因表达报告以显示每个标签的序列,覆盖范围,基因名称和基因组中的位置。还报告了不在库中的新基因标签。这些标签可以作为新标签添加到参考库中。基因名称和基因组中的位置。还报告了不在库中的新基因标签。这些标签可以作为新标签添加到参考库中。基因名称和基因组中的位置。还报告了不在库中的新基因标签。这些标签可以作为新标签添加到参考库中。
十一、de novo NGS大会读
NextGENe包括多种平台特异性装配工具,用于从Illumina,Roche,Life Technologies和Ion Torrent等所有主要NGS平台进行短序列读取的从头组装。NextGENe软件基于Windows的操作提供了一种生物学家友好的无脚本点击操作。 用于Ion Torrent系统的Floton™。
单端或配对数据(包括配对端和配对配对)可用作输入。可以使用多种组装方法,包括de Bruijn组装方法,类似于Velvet组装工具。
NextGENe还包括独特的 Condensation Tool™ ,可在装配前使用,以纠正测序错误和细长读数,从而改善装配效果。这项正在申请专利的技术尚未获得其他项目,如CLC Bio,DNASTAR的NGEN和Lasergene的SeqMan,或者学术工具,如Velvet,ABySS,ALLPATHS&
十二、有针对性的重新测序
NextGENe®软件包括许多有用的工具,可用于生物学家进行靶向重测序项目,包括单核苷酸多态性(SNP)插入/缺失(INDEL)和来自Roche Genome Sequencer FLX / Junior,Illumina GA / HiSeq / MiSeq的测序数据中的大结构DNA重排; Life Technologies Ion Torrent和SOLiD™系统。NextGENe” 基于Windows®的操作消除了CLC Bio,Lasergene的SeqMan和NGEN等程序的复杂性; 以及MAQ和SOAP,Top Hat,BWA和Bowtie等学术软件。生物学家只需输入系统原始读取或BAM文件,选择应用程序并单击开始分析。通过使用附带的条形码分类工具快速解复用合并或条形码的样本,该工具将分离出每个合并的样本用于后续分析。NextGENe的管道工具,AutoRun可用于在无人值守的基础上设置无限数量的连续分析。完成后,结果显示在NextGENe'
专门的重新测序工具包括通过BED文件过滤,感兴趣的区域,删除PCR重复读数,以及与整个基因组对齐或使用一个或多个GenBank(.gb)文件的能力。
NextGENe具有99%以上的精度,INDEL检测高达读取长度的33%; 快速回顾DNA变体,核苷酸和氨基酸; 变异评分; 过滤; 和分析比较。Genome查看器包含指向dbSNP 和NCBI的链接 。
十三、人类身份(法医)分析
它有望使分析更快,更便宜,因为毛细管电泳被消除,并且样品可以用多重标识符(MID)标签进行条形码编码,然后组合在一起进行分析。此外,使用这种测序技术可以增加可用的信息量,例如线粒体,特别适用于降解DNA的分析。比较序列数据而不是电泳结果允许比较样品之间的单核苷酸多态性(SNP)。
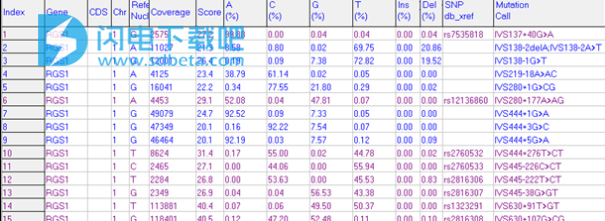
NextGENe是一个非常强大的工具,可用于下一代测序,用于亲子鉴定,刑事法医鉴定和失踪人员鉴定。它能够在几秒钟内准确地对齐数千个读数。内置的条形码分类工具可以轻松使用多路复用样品和NextGENe管道工具,AutoRun允许连续,无人值守的分析。SNP方便地显示在突变报告中,同时计算STR多态性并将结果显示在表达报告中。
十四、使用Ion Torrent PGM和Proton数据进行SNP和INDEL发现
Ion Torrent系统与大多数其他测序系统根本不同。它是一种“后光”技术,因为它不依赖于检测核苷酸掺入的光发射。相反,它使用含有数百万个独立pH计的硅芯片。其基于流动的方法检测在DNA复制中掺入未修饰的核苷酸期间由氢离子释放引起的pH变化。由于这种不同的测序方法,仪器和试剂便宜得多,并且具有独特的错误概况 - 大多数错误是indel而不是替换,特别是在均聚物区域。SoftGenetics创建了Ion Torrent特定算法,以便最好地处理Ion Torrent数据配置文件,当使用配对的结束读取时,将数据质量提高到99.8%以上。
NextGENe将快速解复用合并或条形码样本进行个别分析。包含的管道工具可用于创建多个顺序分析项目。
十五、使用Ion Torrent PGM和Proton数据进行SNP和INDEL发现
Ion Torrent系统与大多数其他测序系统根本不同。它是一种“后光”技术,因为它不依赖于检测核苷酸掺入的光发射。相反,它使用含有数百万个独立pH计的硅芯片。其基于流动的方法检测在DNA复制中掺入未修饰的核苷酸期间由氢离子释放引起的pH变化。由于这种不同的测序方法,仪器和试剂便宜得多,并且具有独特的错误概况 - 大多数错误是indel而不是替换,特别是在均聚物区域。SoftGenetics创建了Ion Torrent特定算法,以便最好地处理Ion Torrent数据配置文件,当使用配对的结束读取时,将数据质量提高到99.8%以上。
NextGENe将快速解复用合并或条形码样本进行个别分析。包含的管道工具可用于创建多个顺序分析项目。
十六、NextGENe AutoRun自动化管道
NextGENe的AutoRun工具允许用户快速设置作业文件,以指定用于分析多个项目的样本文件,参考文件和设置。NextGENe AutoRun工具包括一个作业编辑器,允许在一个步骤中使用点击操作创建作业文件。NextGENe AutoRun扫描作业文件以检查处理作业所需的所有文件的可用性。当文件可用时,NextGENe AutoRun启动NextGENe以开始处理作业。无人值守地连续处理作业。如果某些文件不可用,NextGENe AutoRun将定期以NextGENe AutoRun设置中指定的时间间隔重新检查文件。可以轻松编辑作业文件以添加其他作业或编辑尚未处理的作业。此外,当NextGENe当前正在处理另一组作业时,可以将其他作业添加到队列中。
十七、定制下一代(NGS)测序分析结果
NextGENe软件包括定制所有NGS测序平台数据的分析报告功能,包括Illumina GA,HiSeq,MiSeq,Ion Torrent Proton和PGM以及Roche Junior和其他平台。该工具允许每个设施或实验室仅选择和报告特别感兴趣的分析和质量控制指标。
使用NextGENe软件中的摘要报告工具创建自定义报告。报告选项包括单个PDF中的分析结果和质量控制结果。该工具允许创建自定义标题,包括分析师,审阅者和项目特定注释,包含运行统计信息,详细说明覆盖范围的多个QC图表以及识别失败的扩增子的表格。
可以在处理样品之前或之后设置摘要报告。新的报告功能简化了分析流程,同时提供了所需的灵活性。报告配置可以保存并轻松应用于后续分析项目。报告配置只需指向并单击所需的报告元素即可,
十八、三重和多样本变体比较
这是快速分类数千个突变调用的有用工具,以便找到几十个(甚至更少)候选突变,这些突变可以通过Sanger测序确认并评估其表型影响。来自导入轨道的信息,例如COSMIC,dbNSFP,dbSNP和任何自定义轨道,
有多种选项可用于创建比较:
•基于样本关系,表型和适用的继承模式
•基于每个样本中指定的突变类型
•显示共享变体
•显示不同的变体
十九、注释轨迹
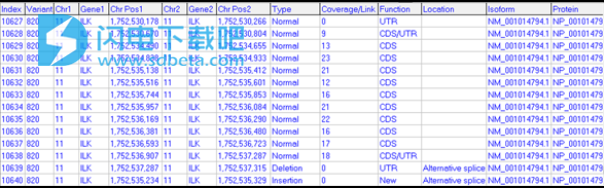
NextGENe中的Track Manager工具允许从外部数据库导入变体注释,可以自动查询变异注释,以便在变异报告(图1)和变体比较报告中使用。可以将诸如dbSNP,COSMIC,1000基因组,dbNSFP和NHLBI GO外显子组测序项目(ESP)等轨道添加到您的项目以及您自己的自定义数据库中。
有四个数据库提供专门支持。有关其中一些的更多信息可以在NextGENe'Help'菜单的'References'对话框中找到。
dbscSNV - 带有dbNSFP的附加数据库 - 功能注释和集合
剪接位点取代的预测
COSMIC (癌症中的体细胞突变目录)
的dbSNP / ClinVar
还有一个Custom Variant Track导入选项,可用于导入上面未具体列出的其他数据库。
1、在查看器中查询曲目
“查询参考曲目”对话框可用于导入或更新对齐项目中的曲目信息。
列出了该项目适用的基因组构建的可用轨迹(图2)。可以选择每个轨道,以便可以在项目的报告中使用它们,也可以取消选择。
2、使用曲目过滤
对于为项目选择的任何轨道,“变化轨道设置”对话框中都有可用的显示和过滤选项(图3)。您可以选择左侧的任何轨道来定义轨道的任何过滤设置,和/或单击“报告显示”以选择要在报告中包含轨道的列。
二十、ThunderBolts™面板分析
当今癌症研究人员面临的主要挑战是能够使用高度准确,简单和低成本的工具快速识别实体肿瘤和血液癌症中发现的特定表型的遗传变异。RainDance ThunderBolts™系统,ThunderBolts NGS基因板的组合,和SoftGeneticsNextGENe®软件可以进行靶向测序,用于研究与癌症疾病具有临床相关性的重要癌基因。该解决方案具有针对每个基因组的预加载分析模板和用于检测不同灵敏度设置(1%和5%)的次要等位基因频率的预定义设置。
NextGENe 2.4.1版引入了AutoRun模板,可以使用各种类型的库准备进行快速项目设置。ThunderBolts癌症面板和ThunderBolts Myeloid面板默认安装。可以将它们加载到NextGENe AutoRun工具中,以快速设置作业以处理多个样本。对于每个小组,NextGENe包含一个具有5%等位基因频率截止的模板和一个“
面板模板为NextGENe中的目标重新排序数据提供了一种更简单的方法。模板用于在创建ngjob文件时指定分析的所有设置。当排序数据可用时,AutoRun工具会检测并处理这些ngjob文件。
二十一、使用独特分子标识符(UMI)进行NEBNext直接分析
处理NEBNext使用NextGENe软件的AutoRun工具可以方便直接数据。AutoRun Tool包含用于单击处理NEBNext Direct数据的内置模板。
模板包括基于UMI去除PCR重复。UMI用于重复去除允许增加等位基因频率准确度以提高变体检测的准确性。NEBNext AutoRun模板只需点击几下即可用于快速设置多个样品的批量分析。
NextGENe NEBNext直接模板通过一系列步骤自动引导样本文件,包括删除PCR重复,格式转换,修剪适配器,与人类基因组对齐和变体调用。满足以下所有条件时,将调用变量:
具有变体的读取百分比大于1.5%
Variant有超过3个读数
总覆盖率超过100次读取
变化的正向/反向平衡比大于0.2(均聚物indels为0.8)
变体在目标区域内
NextGENe软件使用Illumina I2文件中的UMI来识别PCR重复。保持具有最高总分的一对重复项以及独特的配对读数进行处理,同时从进一步处理中移除重复读取。
没有重复删除,区域的许多读取看起来相同,从相同位置开始和结束。重复删除后,可以看到更平铺的读取对齐。可能仍会看到一些看似重复的读取(图2)。由于基于I2 UMI序列进行重复去除,因此仅去除真正的PCR重复。
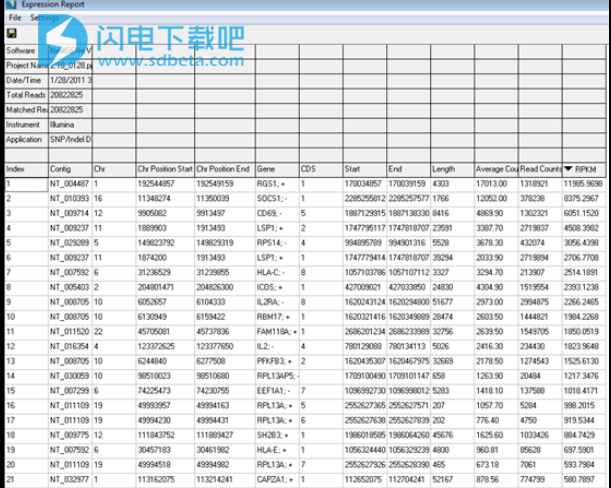
处理完成后,可以在NextGENe Viewer中显示项目(图3)。根据模板设置自动导出若干报告,包括变异报告,若干覆盖曲线报告(使用不同覆盖范围截止报告低覆盖区域)和表达式报告(显示每个目标的读取计数)。
NextGENe的Reference和Track Manager工具允许将可选轨道导入NextGENe软件。然后,可以为每个新项目自动查询这些轨道,包括COSMIC,ClinVar和dbNSFP,为每个变体提供其他信息,以包含在变异报告中。
二十二、使用独特的分子标识符(UMI)去除PCR重复项提供更高的准确性
NextGENe Software的序列操作工具包括使用独特的分子标识符(UMI)删除PCR重复项的功能,例如NEBNext Direct使用的那些或HaloPlex化学品。PCR重复序列的去除提供了确定等位基因频率的准确性,以改善变异体的调用。UMI是随机的碱基序列,用于在文库扩增之前标记DNA的每个分子(片段),这有助于PCR重复的鉴定。
该工具使用Illumina I2文件中的UMI来识别PCR重复。NextGENe识别共享相同UMI的所有读取对,并仅保留要处理的总质量最高的对以及唯一的配对读取,而从进一步处理中删除重复读取。
使用UMI实现重复删除的自动化很容易通过使用NextGENe AutoRun工具创建包含所需分析规范的模板来实现。
通过单击序列操作工具中的“保存”可以保存重复删除设置,如上面的图1所示。然后,可以通过单击“Preprocesses”旁边的“Add”将此设置文件加载到NextGENe AutoRun Tool的作业编辑器中。在为所需的任何其他预处理步骤加载设置文件之后,
然后,只需选择模板,加载示例文件和选择引用,即可使用模板配置项目。
二十三、在瓶子参考样品中使用NIST基因组,通过NextGENe®和GeneticistAssistant®软件验证NGS分析管道
由NIST主办的瓶中基因组(GIAB)联盟提供了参考材料,以促进下一代测序测试适应临床实践。参考材料,基于Coriell的NA12878 DNA,
NextGENe和Geneticist Assistant软件提供完整的用户友好型分析管道,用于从序列发生器的原始数据到策划的变体调用和变体解释的NGS数据。通过专门评估高置信度变体调用,GIAB样本可用于验证最佳分析和报告设置。NextGENe软件提供灵活的变体调用和报告设置,用于过滤变体调用,而Geneticist Assistant NGS Workbench可用于将NextGENe的调用与来自GIAB的样本的已知调用的VCF进行比较。通过这种方式,可以调整设置以消除潜在的假阴性,同时最大限度地减少误报。
NextGENe的变异报告设置可以配置为报告GIAB VCF中的每个位置,以便轻松识别假阴性。
二十四、与Pathway Studio整合的差异基因表达和途径分析
鉴定差异表达的基因产物并了解所涉及基因的生物学途径是了解导致癌症和自身免疫性疾病等疾病的重要机制的关键领域。
PathwayStudio®是Elsevier研发解决方案的产品,利用从数百万篇文章和摘要以及临床试验开发的生物关系知识库,协助研究人员开展这项重要工作。
SoftGenetics的NextGENe软件现在可以直接链接到Elsevier用于Pathway Studio订户的Pathway Studio,从而实现生物系统的无缝挖掘。NextGENe软件的表达比较工具可用于识别差异表达的基因,只需单击一下按钮,NextGENe软件的表达分析结果可以上传到Pathway Studio,实现疾病机制,基因表达等的可视化。这种自动连接可以让研究人员快速访问广泛的知识库,而无需繁琐且容易出错的手动搜索,从而提高实验室效率。 Elsevier研发解决方案。
二十五、NextGENe-Geneticist Assistant Pipeline支持AMP和CAP建议的性能指标报告。
使用NextGENe和Geneticist Assistant管道来支持分子病理学协会(AMP)和美国病理学家学会(CAP)联合推荐的性能指标报告,以进行管道验证(1)。
NextGENe输出一个单独的* _PerformanceMetrics.txt文件,其中包含上面列出的质量指标。指标也会在NextGENe导出的VCF文件中报告。
遗传学家助手允许创建自定义质量度量标准配置文件,允许用户确定要报告的度量标准,设置标记变体的阈值,以及定义遗传学家助手输入文件中可以找到或可以计算度量的位置。默认配置文件是基于AMP / CAP建议内置的。
使用帮助
一、NextGENe主窗口
启动NextGENe时,NextGENe项目向导将在NextGENe主窗口中打开。
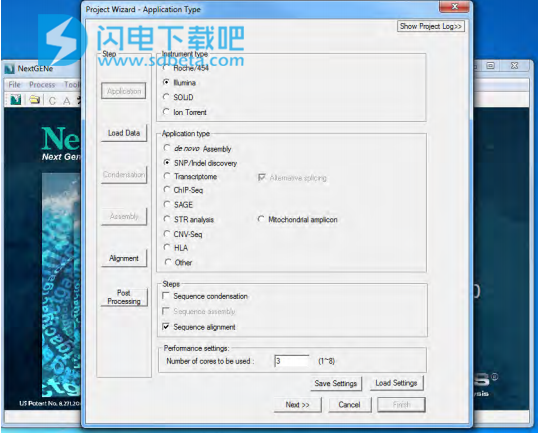
NextGENe主窗口是NextGENe应用程序的起点。 该窗口提供对所有NextGENe功能和系统工具的快速访问。 NextGENe主窗口有三个主要组件 - 标题栏,主菜单和工具栏。
二、标题栏
名称“NextGENe”显示在NextGENe主窗口顶部的标题栏中。 如果已为您的NextGENe实例启用了用户管理,则您的用户名也会显示在标题栏的括号中。
您正在运行的NextGENe版本不会显示在标题栏中。
您必须使用主菜单中的帮助>关于选项来确定版本号:请参阅下面的“主菜单”。
三、主菜单
主菜单以标准Windows菜单格式设置,菜单命令分组到菜单栏中的菜单(文件,处理,工具和帮助)。 其中一些菜单命令可在应用程序的其他区域中使用。
四、在NextGENe中管理用户
用户是登录NextGENe的人,无论是添加和查看内容,还是仅以只读容量使用NextGENe。 如果您是NextGENe的管理员用户,那么您负责管理NextGENe实例的所有其他用户。 管理NextGENe的用户包括添加新用户,编辑现有用户和删除用户。 您还可以在日志文件中查看NextGENe用户的活动(登录或注销NextGENe)。
1)在NextGENe中管理用户
1.在NextGENe主菜单上,单击“帮助”>“用户管理”>“管理设置”。
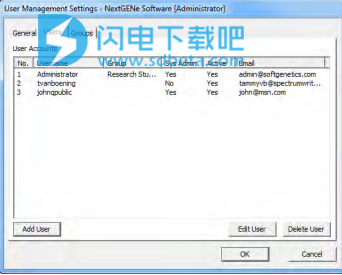
将打开“用户管理设置”对话框。 “常规”选项卡是打开的选项卡。 请参见第38页的图1-18。
2)(可选)要在日志文件中查看NextGENe用户的活动(登录或注销NextGENe),请单击“查看日志”。
用户管理日志文件将在屏幕上打开。 该文件列出了NextGENe用户的登录和注销活动,如果适用,还列出了您的Geneticist Assistant用户的所有活动。 您可以单击“保存到文件”以使用您选择的名称和位置保存日志文件。
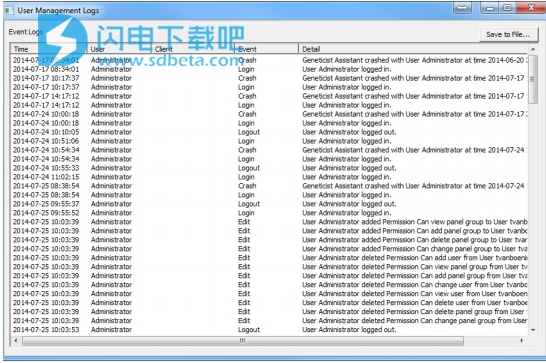
3)单击“用户”选项卡将其打开。
该选项卡列出了已为NextGENeinstance配置的所有用户帐户,以及已为您的Geneticist Assistant实例配置的任何用户帐户(如果适用)。