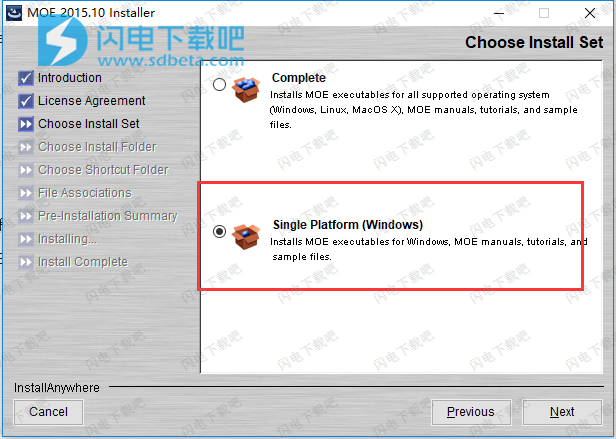
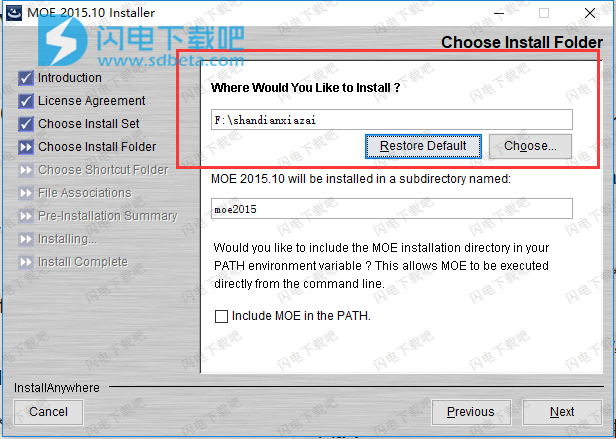
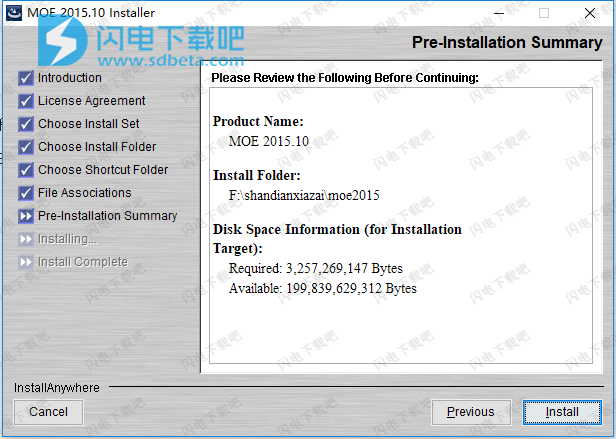
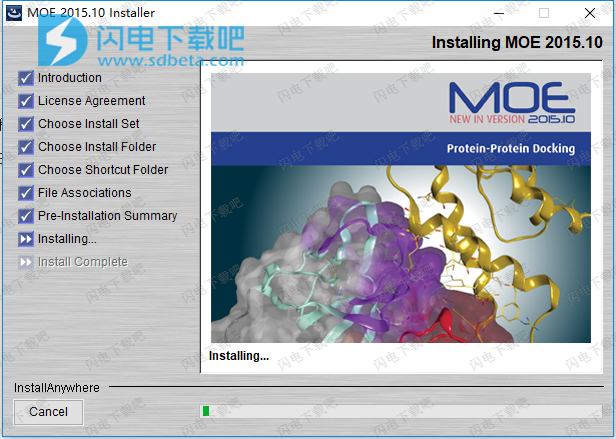
生长,连接和替换[Grimshaw 2010]配体支架(有或没有受体)。药物化学转化对依赖于使用标准草图产生的反应转化(.rxn)的分子进行小的等量变化。 CCG提供了170多个转换的数据库。如果可用,优化活性位点中的坐标(柔性受体)并计算结合分数,同时保留药效团特征。应用2D和3D描述符过滤器,QSAR和指纹模型。
药物化学应用
CCG在为铅生成和优化领域的药物化学家创建和部署解决方案方面拥有十多年的经验。分子操作环境(MOE)已被许多顶级药物研究公司采用,用于大规模药物化学部署。许多中小型制药公司也将MOE作为其主要的药物化学建模平台,以加速新疗法的开发工作。
适用于药物化学家和计算科学家的协调平台
多个不同发现项目组之间的无缝通信
易于与内部数据库,服务器和管道工作流系统集成
CCG(与大型制药公司合作)开发了一个简化的界面,用于主动站点可视化和配体优化。按钮栏提供结构准备,活性位点分析,分子特性/结合亲和力计算,潜在R组方向(替代机会)和活性位点配体优化的应用。使用3D构建器或使用2D草绘器修改配体。测量距离,角度和二面角轮廓。可视化和修改对齐的复合物,打开/关闭蛋白质,浏览对接姿势,药效团命中并使用系统管理器调整渲染
1、MOEsaic:用于配体分析的Web应用程序
MOEsaic是一种基于浏览器的应用程序,用于分析药物发现项目中的一系列小分子化学结构和相关属性数据。对齐分子以促进成对比较。进行子结构和相似性搜索。进行匹配分子对(MMP)分析。使用内置化学草图定义具有确定支架的R-基团。检测活动悬崖和生物电子等排。通过属性图和应用的过滤器可视化数据。使用文本和图像设计虚拟结构和文档结果。
2、可视化和分析非键合相互作用
使用扩展Hückel理论(EHT)可视化和分析配体 - 受体相互作用,如氢键,包括CH..O相互作用,卤素键,硫 - 氧相互作用,质子 - 和阳离子 - π相互作用。 EHT更准确地计算相互作用强度并考虑电子撤回和共振效应。
3、蛋白质 - 配体相互作用图
MOE - 药物化学应用自动生成与配体或一系列配体相互作用的活性位点残基的2D图[Clark 2007]。在2D中可视化关键的相互作用,如氢键,盐桥,疏水相互作用,阳离子-π,硫-LP和卤素键。使用描绘的空间轮廓识别配体取代的潜在位置。可视化溶剂暴露的配体原子和具有强疏水相互作用的残基。浏览化学系列或受体家族系列,以确定选择性分析的保守或非保守相互作用。
4、曲面和地图
构建由属性着色的分子表面,以定义和表征活性位点拓扑并识别配体取代机会。预测基于知识的非键合接触偏好或使用非线性Poisson-Boltzmann方程计算静电图以识别高值疏水区域和极性热点。使用3D-RISM计算水密度和结合去溶剂化惩罚图,3D-RISM是基于液体密度泛函理论的溶剂化的第一原理理论。检测由相关性和空化效应产生的结合位点的非明显疏水区域,以优先化配体修饰。
5、构象搜索和分析
探索配体构象空间,以获得有关生物活性构象和分子内相互作用的见解。使用LowModeMD [Labute 2010]通过执行快速隐式振动分析和短分子动力学模拟来生成大环和多组分系统的构象(例如,显式水或反离子)。
6、多分子的灵活比对
执行已知和推定配体的3D比对(或叠加)以确定生物活性的结构要求 - 特别适用于基于配体的药物设计方案,因为对齐的基团可能对于确定生物活性构象是重要的。使用全原子灵活对齐程序[Labute 2001],该程序结合了力场和基于形状和药效团特征的高斯描述的3D相似性函数,以产生小分子集合的可能对齐的集合。
8、脚手架更换,成长和片段链接
生长配体,连接片段并替换支架[Grimshaw 2010],用于快速后续化合物,包括创新的线性,环状或融合支架排列。在(柔性)活性位点中细化新结构,同时保持重要的药效团相互作用并计算预测的结合亲和力。使用药物化学变换通过对配体进行小的等量变化来探索局部SAR。 CCG使用从化学文献中提取的规则提供170多个功能组,同源和异构转换的数据库。可以使用标准2D草绘器添加新的转换。
9、Pharmacophore Discovery MOE - 药物化学应用
MOE包含业界领先的药效团发现应用套件,用于基于片段,配体和结构的设计项目。药效团建模是生成和使用3D信息以搜索新型活性化合物的有力手段,特别是在没有可用的受体几何形状时。药效团方法使用广义分子识别表示和几何约束来绕过2D方法的结构或化学类偏差。
使用交互式编辑器从分子比对或受体结构构建3D查询。执行构象数据库的虚拟屏幕以确定候选活性化合物。使用SMARTS化学模式(针对特定组)和/或表达定制药效团特征。通过使用包括,排除和外部体积的球体结合来限制形状(受体或配体)。使用原子上的方向向量约束或要素上的部分匹配来优化查询。
10、分子描述符计算
超过400种2D和3D分子描述符,包括拓扑指数,结构键,E-状态指数,物理性质,拓扑极性表面积(TPSA)和CCG的VSA描述符[Labute 2003],广泛适用于生物活性和ADME财产预测。应用基于扩展Hückel的描述符(例如LogP,LogD和摩尔折射率)来计算分子属性。计算小分子的pKa和pKb,并确定在给定pH下配体质子化状态的群体。使用描述符进行分类,聚类,过滤和预测模型构建。使用MOE的内置科学矢量语言添加自定义描述符。
11、Virtual Library Builder
通过基于反应的Combinatorial Library Builder枚举化合物库。使用商业或定制的内部试剂作为反应引擎的输入。进行简单的酯化反应或多组分Ugi型或Groebke-Blackburn-Bienyame反应。使用标准2D草绘器指定反应或多个同时反应步骤。自动筛选与靶标或药效团模型的化学相似性的反应产物。使用化学描述符或Lipinski的五级规则过滤结果以获得药物相似性。通过应用QSAR或药效团模型计算聚焦库。#p#分页标题#e#
生物制剂应用
MOE拥有一套全面的计算机辅助生物制剂设计应用程序,包括面向工作流程的界面,可简化可视化和分析。计算方法有助于识别和优先化潜在的诱变实验,用于性质调节,例如溶解度,亲和力和稳定性。
1、全蛋白和界面可视化和分析
MOE - 表面贴片可视化蛋白质:蛋白质界面区域的非键合相互作用(阳离子-π,氢键,空间碰撞等...)。创建分子表面并分析表面特性,如疏水2、性和静电势。
分析表面贴片以了解局部疏水性和极性。比较多个结构以了解亲和力和结构可变性的差异。突出潜在的反应位点进行氧化和脱酰胺。使用基于知识的潜力对热点进行可视化和排序,并评估非线性Poisson-Boltzmann方程以评估静电偏好,以便合理化相互作用和潜在的诱变位点。
3、蛋白质 - 蛋白质对接预测蛋白质 - 蛋白质结合姿势。
使用粗粒珠模型结合快速傅里叶变换(FFT)生成高质量的对接蛋白质结构,然后进行所有原子最小化。通过使用基于知识的刚体对接来聚焦采样空间。自动检测抗体CDR位点以限制搜索空间。生成并分析蛋白质 - 蛋白质相互作用指纹以确定涉及结合的关键残基。
4、抗体和融合蛋白模型
通过从一个或多个模板组装实验确定的骨架结构的结构域片段,从氨基酸序列构建3D抗体结构或融合蛋白(包括多结构域模型)。使用专门的抗体建模方案[Almagro 2011]。为基于知识的循环建模指定可自定义的循环字典。使用包括MM / GBVI的各种评分函数对同源性模型进行评分。在结构模板中包括环境单位,例如scFv,Fc或抗原片段,用于诱导拟合。
5、蛋白质贴片
可视化高价值的疏水性和带电荷蛋白质贴片,以合理化表面特性并评估易聚集区域。在QSAR和QSPR模型中应用蛋白质贴片描述符,用于预测和调节蛋白质特性,如溶解度和粘度。使用蛋白质贴片检测潜在的结合位点或绘制表位。
6、蛋白质特性
为QSPR建模计算一组全面的基于序列和结构的物理特性,如pI,zeta电位,迁移率,偶极矩等。将预测的性质与初步实验数据结合使用,以使给定pH下的稳定性和聚集合理化。计算突变体集合的属性,以识别和预测相对规模的物理属性趋势。
7、蛋白质工程
MOE - 蛋白质工程通过统一的蛋白质工程应用探索和比较突变系列与野生型。进行丙氨酸扫描以系统地探索亲和力。通过半胱氨酸扫描识别二硫键桥接机会,评估蛋白质稳定性并优化不稳定区域。通过残留扫描合理化并执行单点或多点突变,以评估和推进候选铅。使用序列设计搜索所有可能的多个突变以确定最佳序列。基于单核苷酸多态性,使用抗性扫描,轻松识别易于自然突变的残留物。使用分子动力学或LowModeMD自动生成集合以估计集合平均属性。
8、突变和旋转探针
使用MOE的Rotamer Explorer进行单点突变并发现氨基酸可及性。使用基于能量的评分函数预测3D蛋白质结构和候选旋转异构体中氨基酸突变的结构。使用MOE的图形界面可视化和分析新的交互和属性。
9、序列分析器和编辑器
使用集成的序列编辑器在残留级别可视化和修改结构。通过切割和粘贴残基进行环路移植或构建序列,蛋白质,DNA或PTM结构来编辑序列信息。使用Rotamer Explorer突变残基并评估旋转异构体。使用CCG独特的技术找到蛋白质序列和结构叠加的最佳比对。自动注释抗体并应用对齐约束以实现最佳叠加。使用序列编辑器以交互方式调整对齐。通过功能,序列相似性或结构接近度动态地着色残基。
10、先进的分子模拟
应用简化的结构准备和优化过程。运行分子动力学(MOE或NAMD)来评估环路或溶剂中的稳定性和总运动。运行LowModeMD for generati